【PyMOL】どこが違うのかな?
今日は有給なので、久しぶりに生存報告です。
生きてます!
さて、我が家の子供たち(自分も含む)はサイゼリヤが大好き。間違い探しも大好き。
ということで、今回は「タンパク質のアミノ酸、違うのどーこだ?」をやってみましょう。
きっかけ
PyMOLで相同性を見てみました。
— おじさん (@snowborderjack) 2022年8月1日
python使ってもっとスマートに仕事したいなー。
はてなブログに投稿しました #はてなブログ
PyMOLで相同性を確認してみた - 或る化みす途のブログhttps://t.co/1iwquWR9pk
巷で話題の"おじさん"さんのブログです。同一低分子化合物とJAK1, JAK2との複合体X線結晶構造(PDB_ID: 6BBU, 6BBV)を重ね合わせて「違うのどこだ?」をする方法を紹介されています(詳しく書かれているので是非ご覧ください。その他にもPyMOLの使い方について書かれた記事もあり、とても参考になります!)。
その中にこんなコメントがありました↓
このsequenceを見ながら一致してない残基を見つけてやればいいわけです。
(私のcoding能力では自動抽出がまだできませんごめんなさい)
結構需要のある作業だと思われます。便利ツールがPyMOL Wikiに転がっているので紹介しますね。場合により上手く動かないこともある?みたいですが、私の場合はWindows, Mac, centos, ubuntuで動作しています。それではスタートです!
Color By Mutations (from PyMOL Wiki)
今回紹介するスクリプトです。
上記のリンクを踏むと、こんなページに辿り着きます。
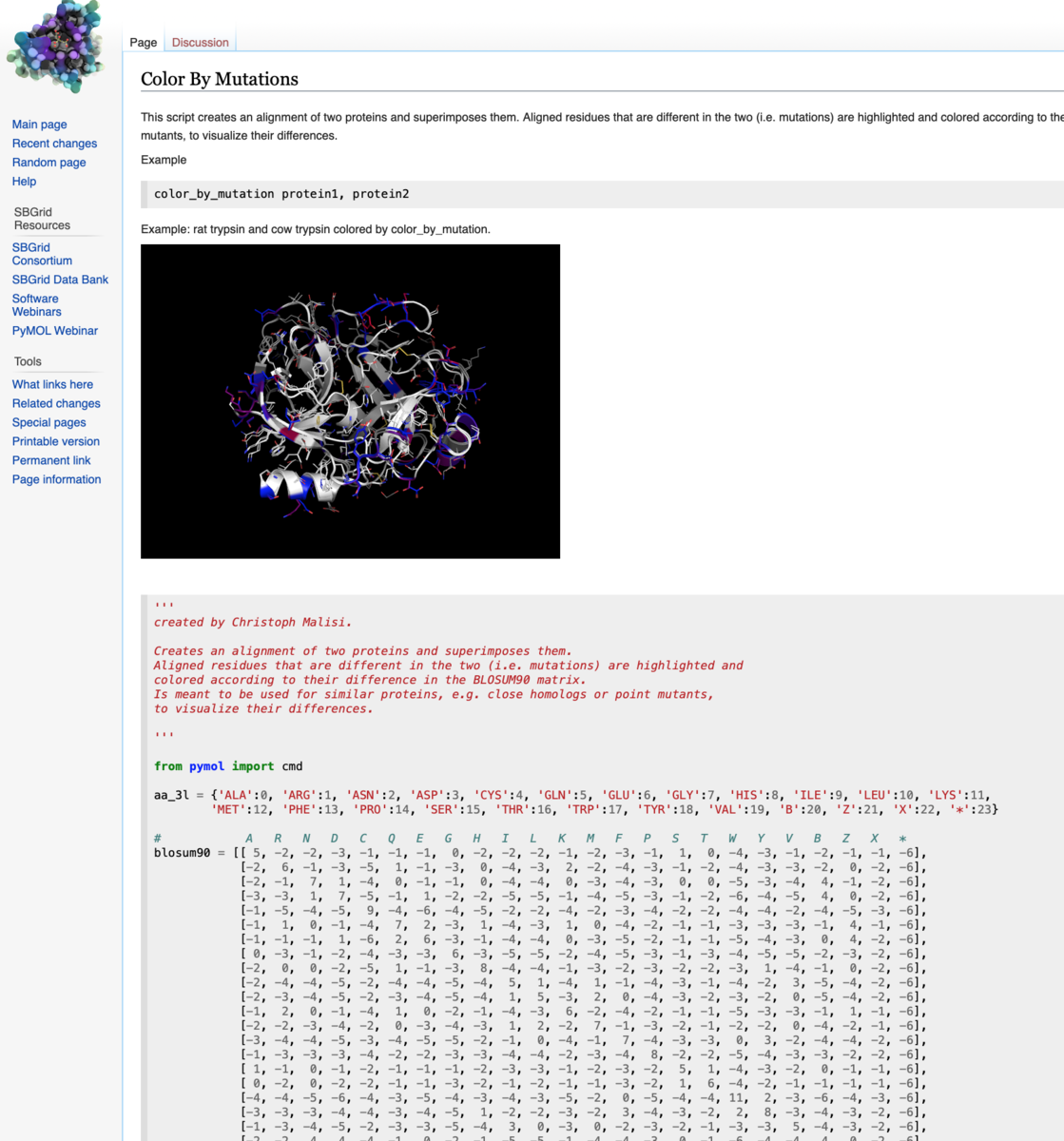
ものによっては、右上にpython scriptのダウンロードリンクが貼ってあるのですが、Color By Mutationsにはないので、グレー背景部分のコードをコピーして自分のマシンに保存しちゃいましょう!
これで下準備は完了なので、少しPyMOLをいじってみましょう。今回はMacでやってみます。使ってるPyMOLはbrewでインストールしたもので、ちょっと古いやつです。
color_by_mutation を使えるようにする
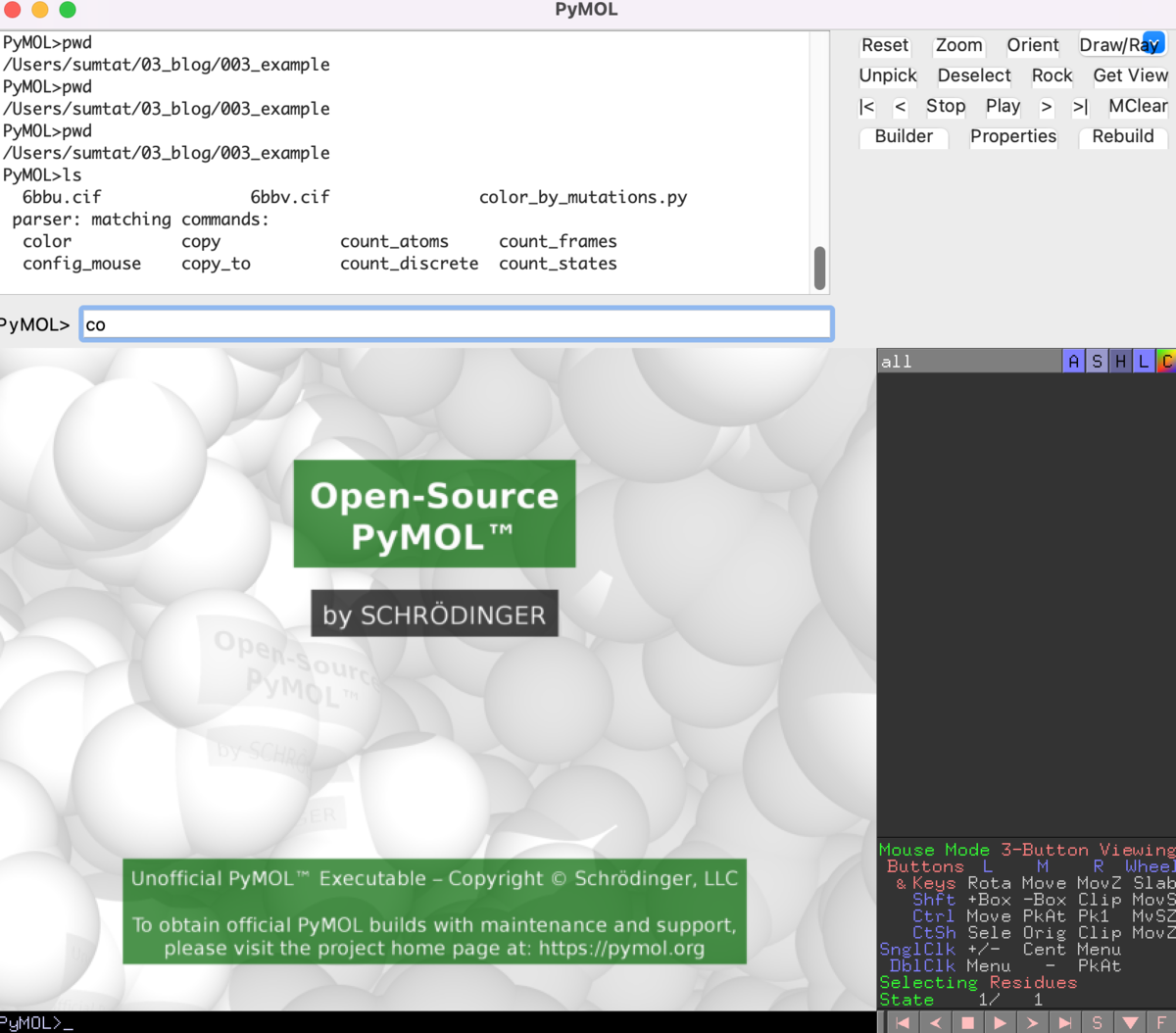
私の場合、先ほど保存したpython scriptは"color_by_mutations.py"という名前で、
"/Users/sumtat/03_blog/003_example"という場所に保存してあります。この時点では
"coと入力後、Tabキー"を押しても、color_by_mutationが候補の中にないことが上記のキャプチャで確認できます。"color_by_mutations.py"の中にあるcolor_by_mutation関数を使えるようにするために、次のコマンドを実行します。
"run color_by_mutations.py"
すると"coと入力後、Tabキー"を押せば、color_by_mutation関数が候補として表示されますね!(この方法だと、PyMOLを起動後、毎回同じ作業が必要)
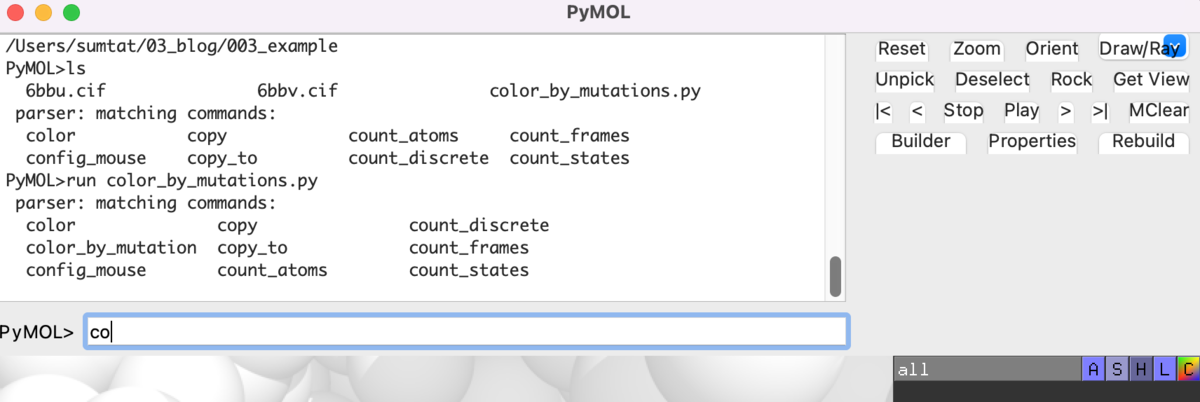
別法としてはメニューバーにてPlugin > Plugin Maneger > Install New Plugin > Choose file... と進んで、該当のpython scriptを選べばOKです(これだと、毎回起動時に勝手によみこんでくれます)。
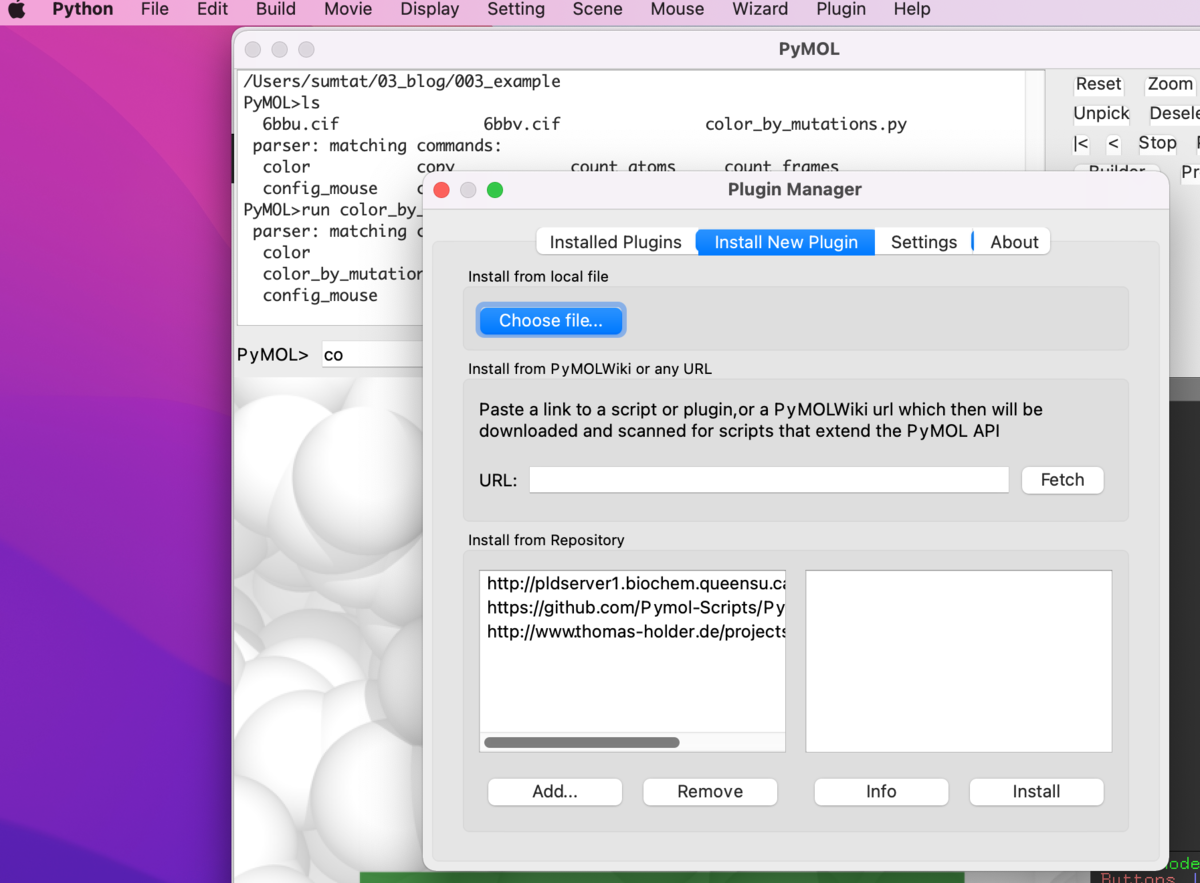
うまくいかない場合は、手動で該当のpython scriptをstartupフォルダに入れてあげればOKです。場所はInstalled PluginsのInfo(何かしら入っていると思うので)をみて、あたりをつけて下さい。↓はapbs_toolsという既存プラグインの場所をGUIで表示させている例です。
さて、color_by_mutation関数を使ってみましょう!
まずは"おじさん"さんのブログに倣って、6BBU, 6BBVを読み込んでおきます。続いて今回の主役"color_by_mutation"関数を動かします。
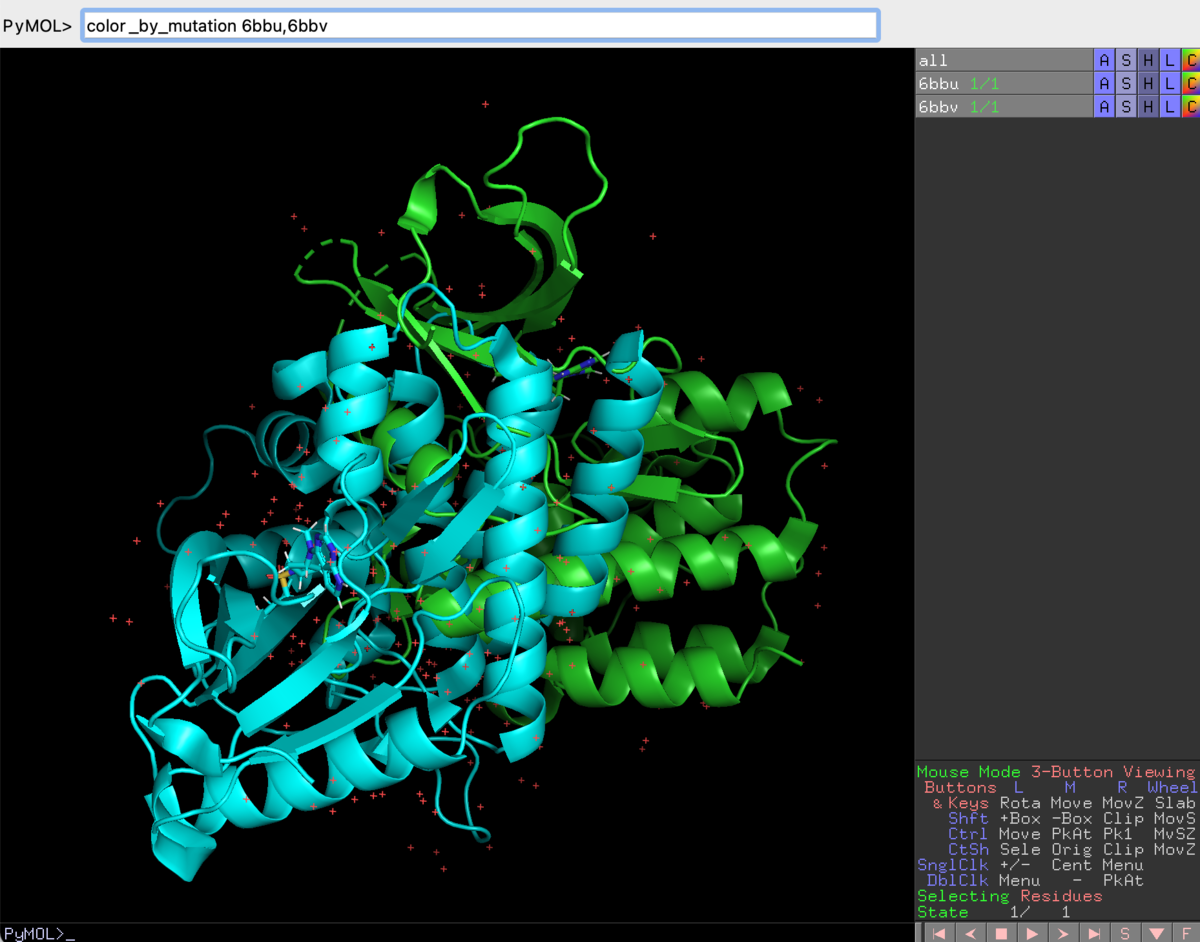
"color_by_mutation 6bbu, 6bbv"を実行すると、こうなる↓
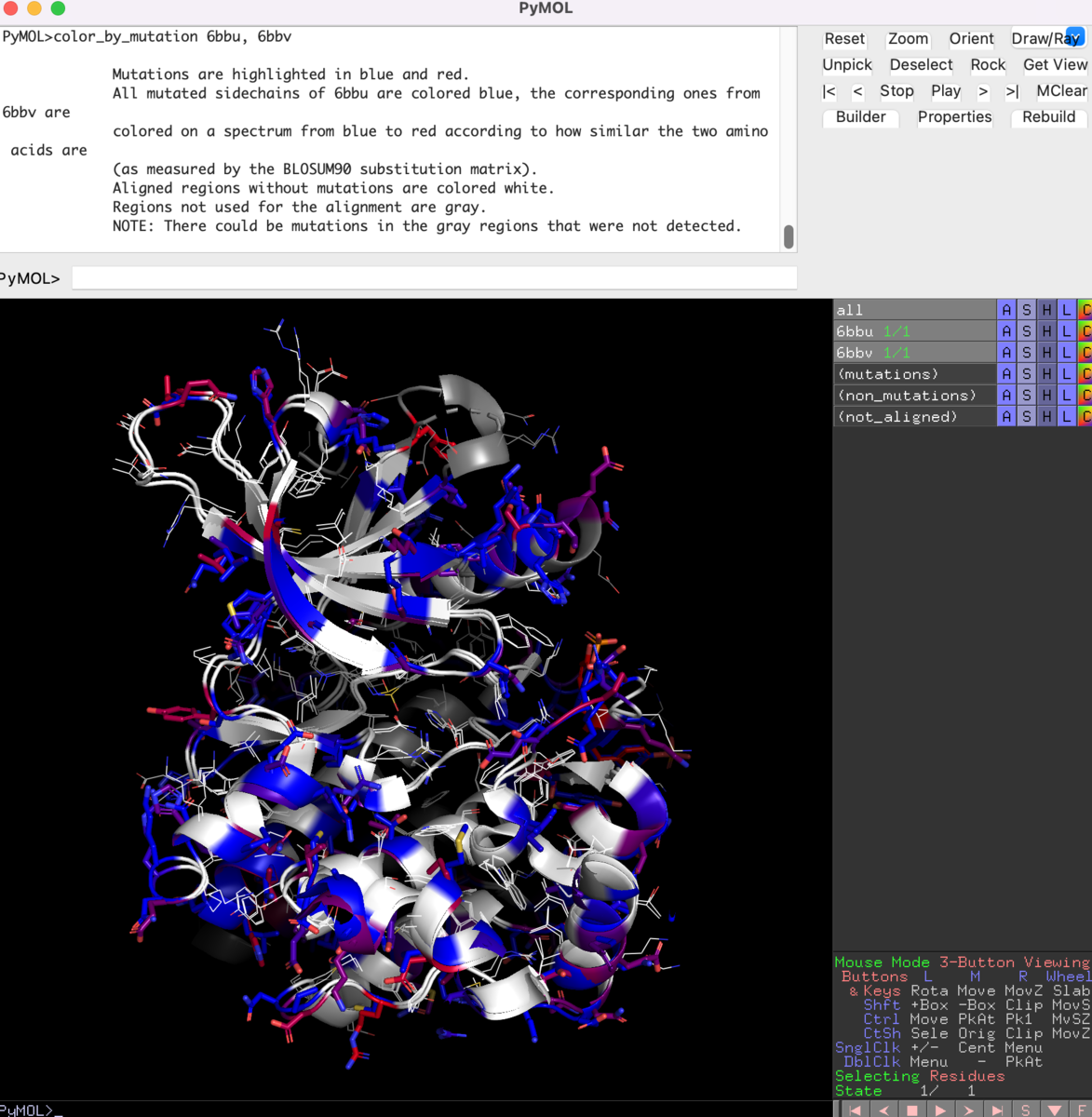
アミノ酸残基の異なるところに色がついて、stick表示になります。6bbuの方は青で、6bbvの方は、対応するアミノ酸の類似度(BLOSUM90パラメータにて算出)により青(似ている)〜赤(似ていない)のグラデーション表示されます。この状態だと、配列アライメントの情報を保持していないので、表示用にもう再度alignして、低分子リガンドも表示などなどしておきまひょか。だいたいこんな感じかな?
-
align 6bbu, 6bbv, cycles=0, object=aln
-
select ar7, byres resn d7d around 7
- hide lines
- hide sticks
-
show sticks, resn d7d
-
show sticks, ar7 and mutations

今日は有給で、家では論文アクセスできないんで詳細はわからんですが、p-loopの動きがかわってくるんですかねー。
ということで、PyMOLは結構便利ですよー。
それではまた次回!

