Neural Network Consoleで活性値予測をしてみた
SONYからNeural Network Consoleというのが公開されましたね(以下NNC)。
これの良し悪しを議論できるほどの知識は私にはないです。でもGUIツールということで、とりあえず使ってよう!という試みです。セットアップの方法などは公式に書かれていますので、そちらを参考にしてください。
さて、なにをやってみようかなと考えたのですが、私は仕事柄、画像認識をあまり活用できる身近な内容があまり思いつかなかったので、数値(阻害活性値)の予測をしてみることにしました*1
チュートリアルには画像の分類(MNIST)が書かれているのですが、画像以外の形式を入力にする例はあまり紹介されていません。私はこの記事を参考にしました。
この内容に従えば、とりあえず動きます。ざっくりというと、
の2つがあればいい。という内容です。
データのcsv

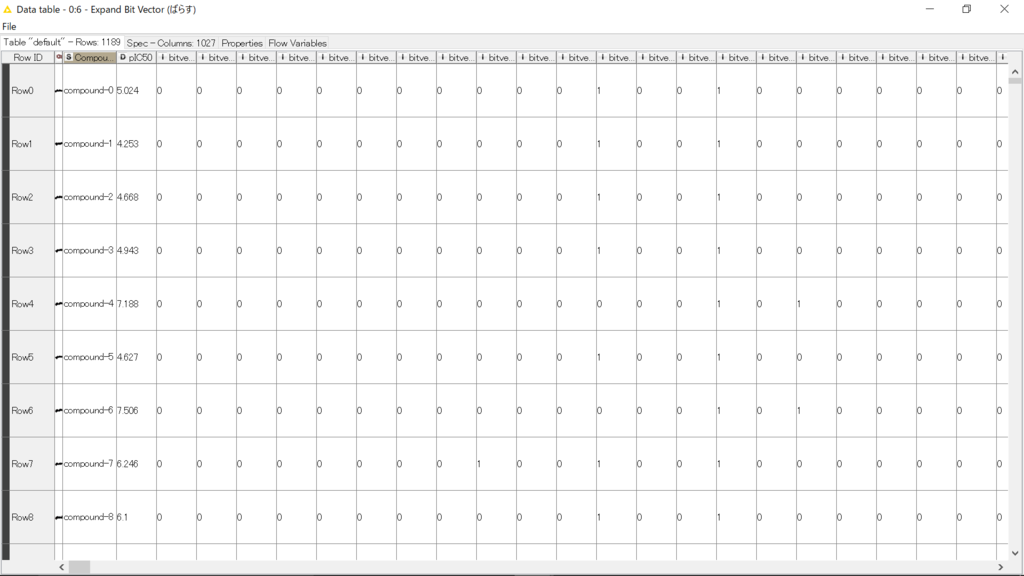
今回の場合は、ただの0,1が1024列並んだcsvが化合物ごとに必要ということです。上記はcompound-0についてのもの。これが1つの場所にたくさんある状態が必要です。

これ、誰でも簡単に用意できるものなのでしょうか?
KNIMEを使えば、結構簡単ですよ。過去記事の内容ですべて網羅できますので、あとでちょっと解説します。
データセットのcsv
これは何を指すかというと
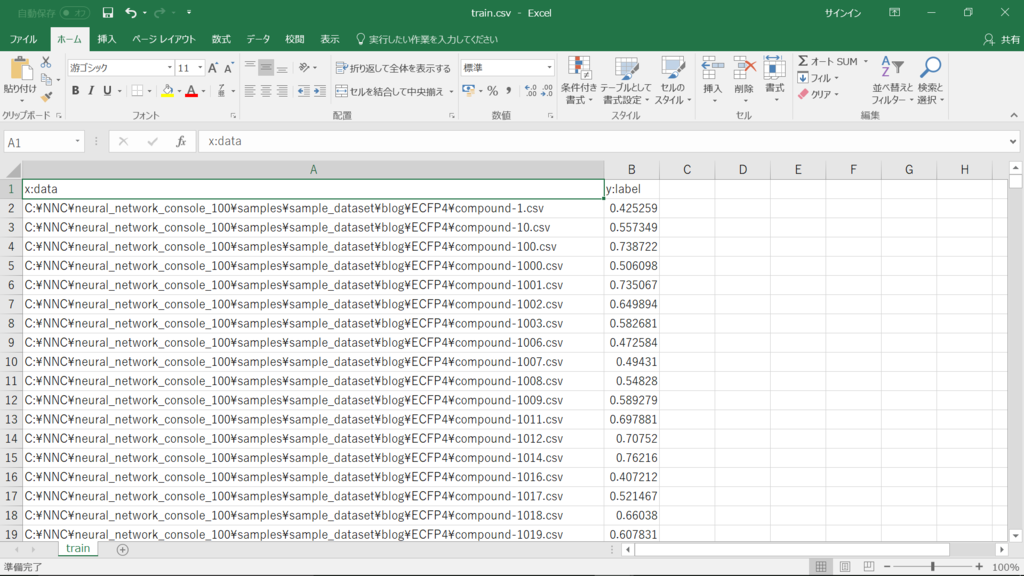
ラベルとは、今回の場合「正解の数値」ですし、分類をしたいならred, blue, greenなどの「正解の種別」に該当するものです。今回は活性値(-1~1にして欲しいみないなので、pIC50を10で割りました)。このリストを作る時もKNIMEで簡単に作れます。
ここまでで紹介した2種の準備物を作成するフローをキャプチャしました。
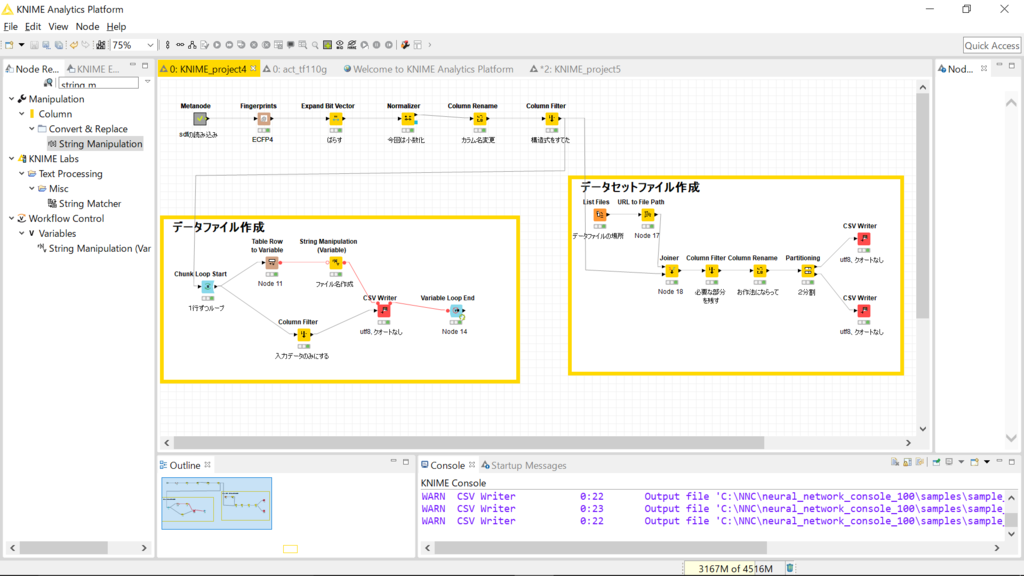
ちょっと細かいですね。
過去記事のリンクを活用しながら、ざっくりと解説します。
fingerprint関連
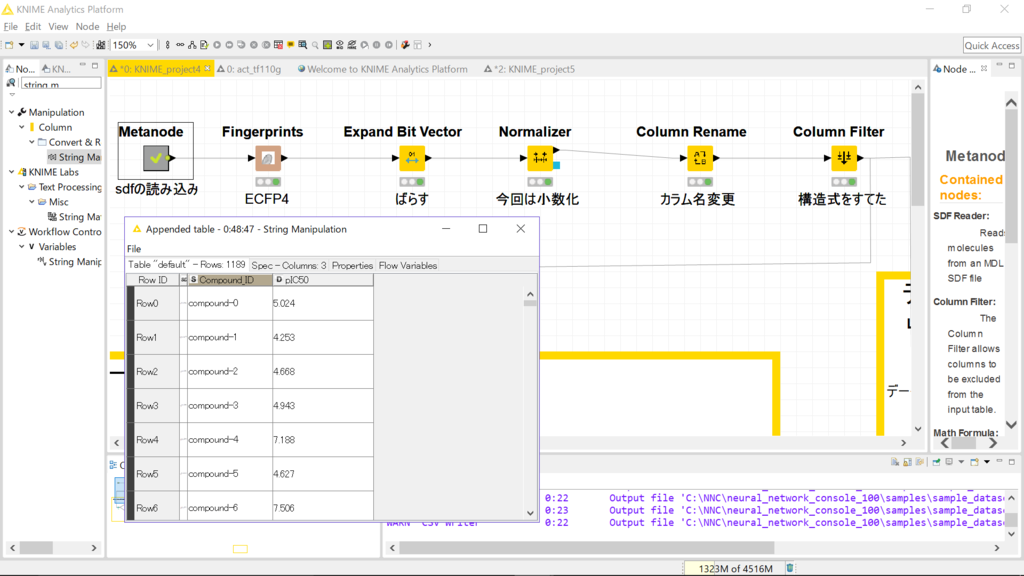
sdfを読んだ後です。化合物のIDとpIC50が入ってます。構造も入ってますが、自分のデータなので見えないようにしてあります。*2
これにFingerprintsというノードを繋げます。
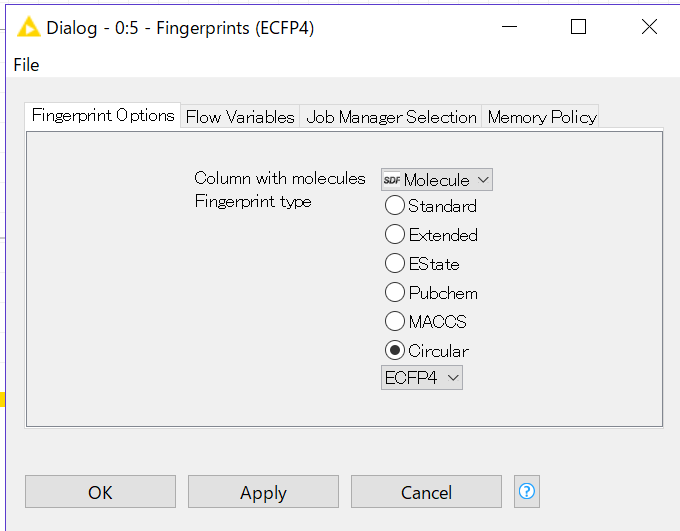
設定画面はこんな感じです。特に意味はありませんが、Circular-ECFP4を今回は選びました。これを実行した後にExpand Bit Vectorというノードをつなげると0,1のビットが1024個、個別のカラムに分割されます。
この内容のpIC50をnormalizeします。
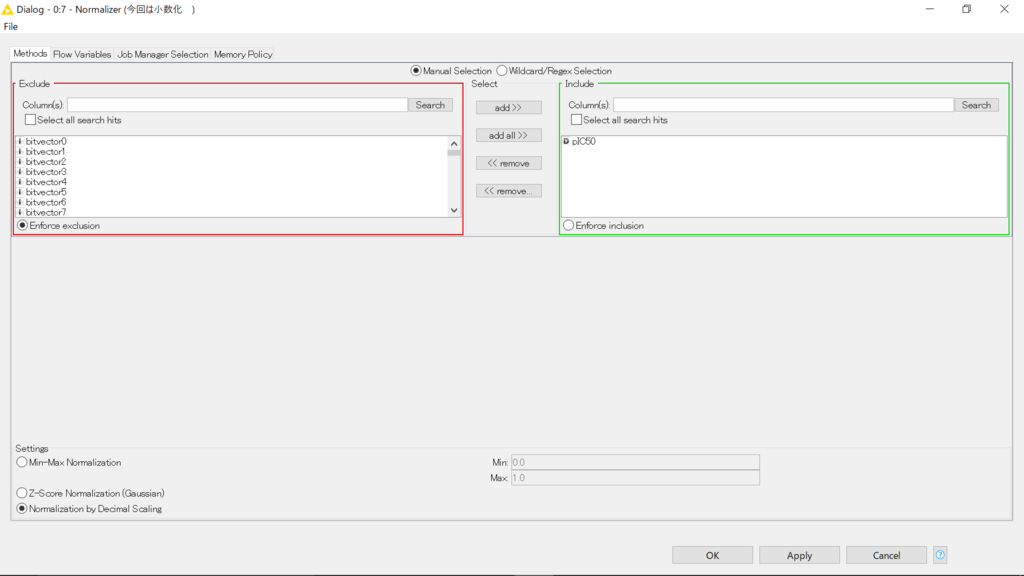
normalizerの設定画面です。乱暴かなと思いましたが、単純に小数化しました(キャプチャ左下)。
続くColumn RenameでpIC50というカラム名をpIC50_normalに変更し、Column Filterで構造式のカラムを捨てました(fingerprintを出したので、もういらない)。
データファイル作成
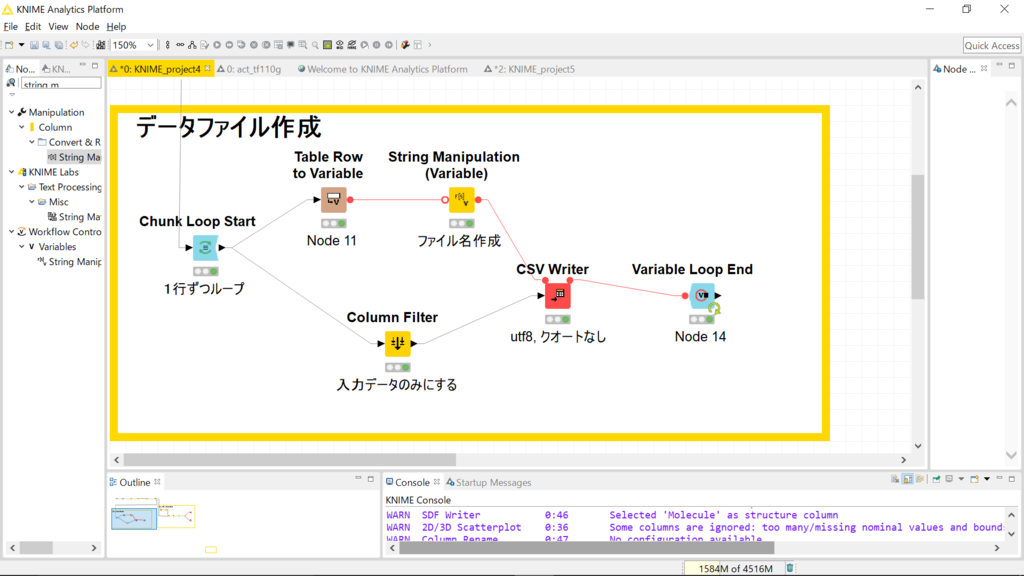
Loopを使って1行ずつ読み込み、csvに書き出します。分岐上側では、変数化された化合物のIDを「所望の場所+化合物のID+拡張子」になるようにしています。分岐下側では化合物IDとpIC50_mormalを捨てて、fingerprintのみ(学習に使う入力値のみ)になるようにfilterしています。
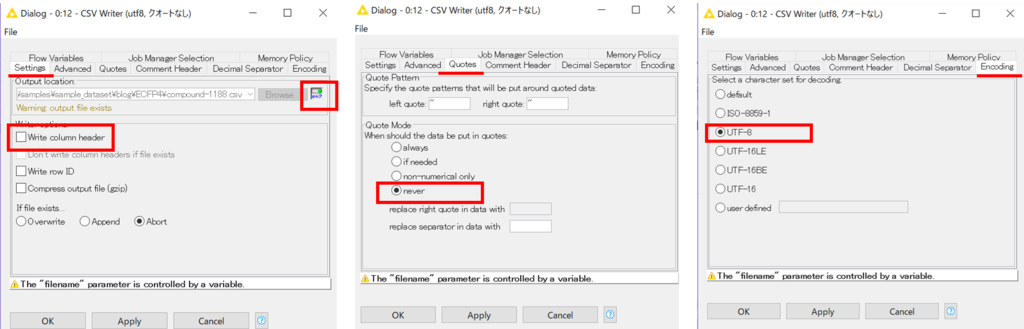
上側分岐からきた変数をファイル名として、csvファイルを書き出します。書き出し時には、カラムヘッダは入れません。また、クオートもつけないように指定しました。文字コードはUTF-8としました。各種タブにて設定可能です。
ここまでで、冒頭のファイルいっぱい状態が出来上がりです。
データセット作成
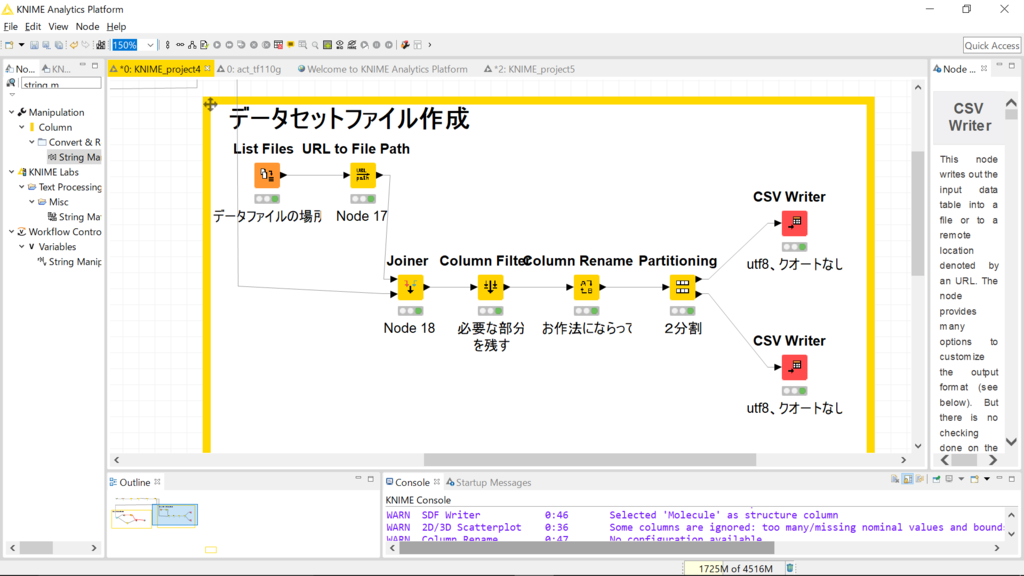
データをたくさん格納した場所をList Filesで指定します。続けてURL to File Pathを繋ぎます。するとFile nameというカラムができますが、これは今回の例では化合物のIDとして作成してあります。ですので、元のデータの化合物IDとfile nameでアノテーション(joiner)したあとに、いらない項目を除けばおおむね完成です。
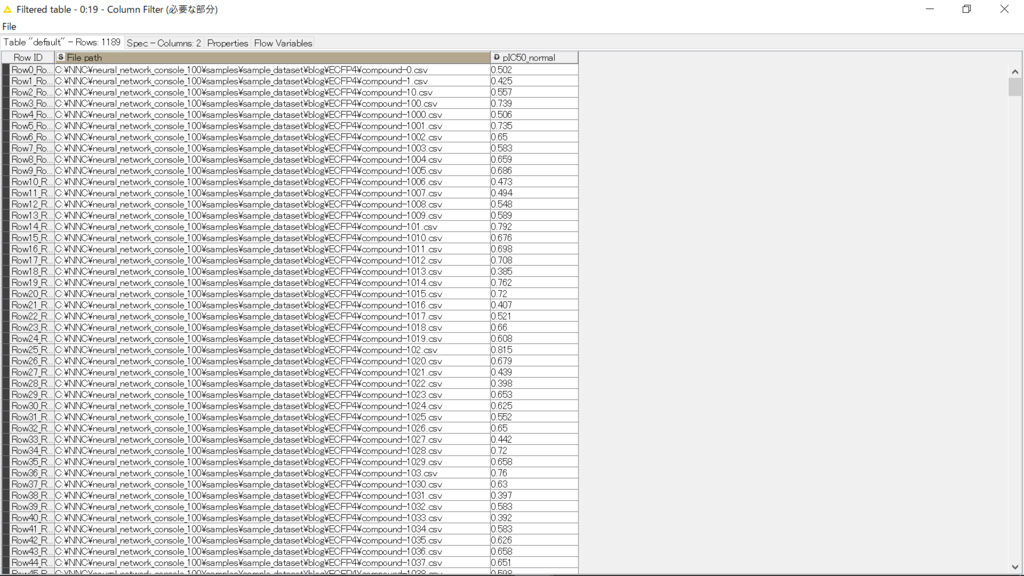
データの場所と、活性値(正解の値)だけのテーブルができています。NNCのお作法に従ってカラム名は
- ファイルの場所→【x:data】
- 正解値→【y:label】
に変更します。このtableを学習用と評価用に分割すれば準備完了です。partitioningノードでは様々な方法でデータを分割できます。今回はランダムに80:20に分割しました。この分け方とかも、deeplearningには重要なんだと思いますが、今回はあまり気にしていません。
最後にそれぞれをcsvとして書き出しました。
NNCの画面
とりあえず、1024→100→1という感じにしました。最適化法はデフォルトのままでadam、lossはsquared errorにしました。勉強を始めようとしているところなので、意味ねーよこんなのという内容でしたら申し訳ありません。いろいろ教えてくださる方がいると助かります。
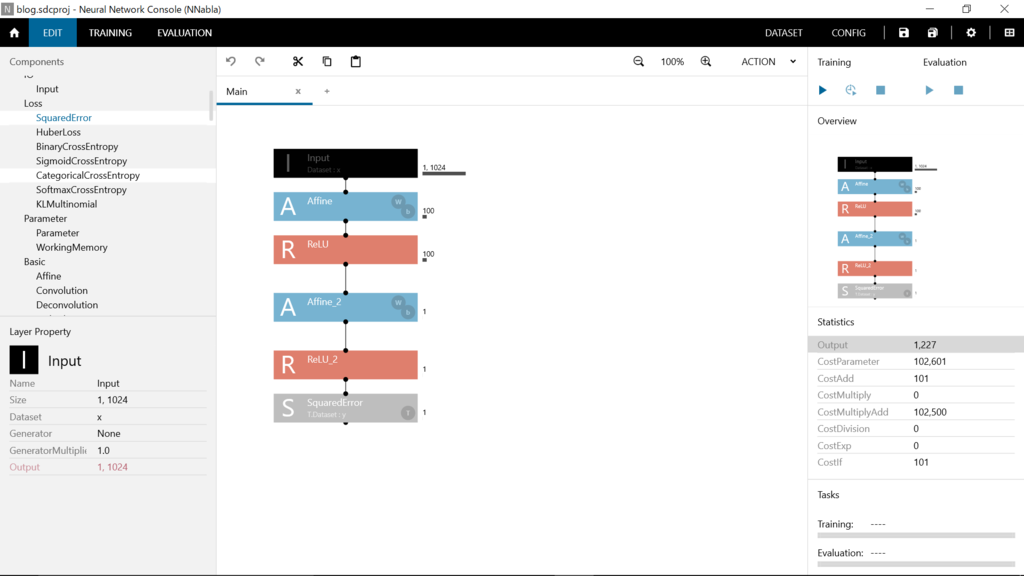
これで学習するとこんな感じのようです。

評価結果はこんな感じ。
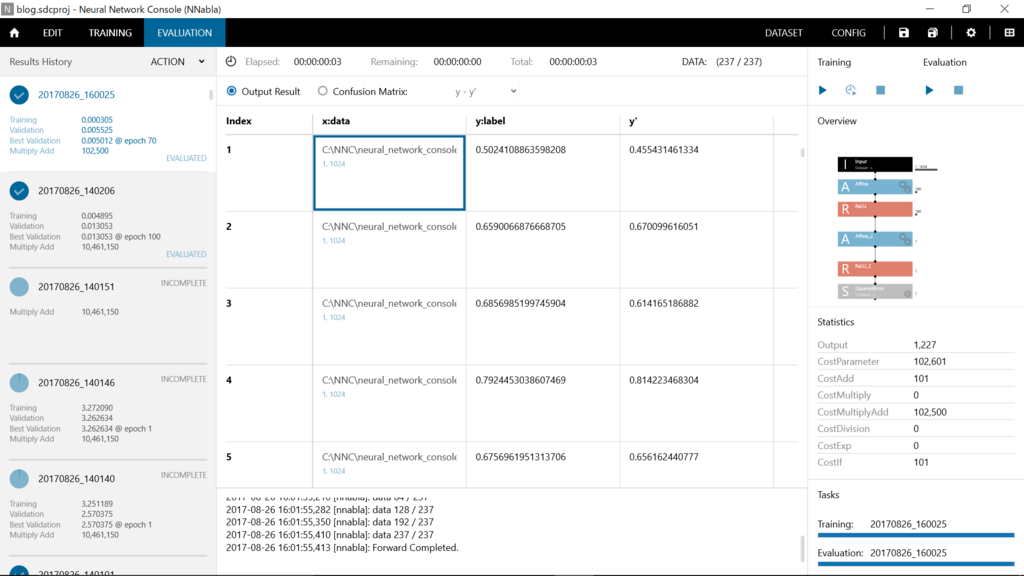
y:labelが正解(pIC50を10で割ったやつ)、y'が予測値になります。
この画面で、右クリックすると内容をcsvで書き出すことも可能です。
書き出したcsvをKNIME上でグラフにしてみました。
ファイルを読み込んで、2D/3DScatterplotにつなぎます。
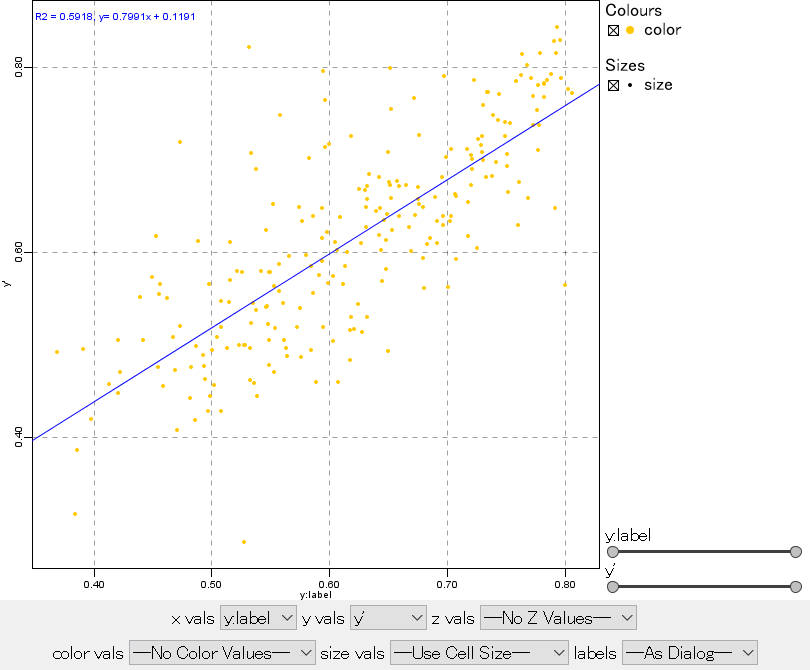
ちょっと見にくいですが、横軸がy:label(正解)・縦軸がy'(予測値)です。r^2 = 0.59となっています。なかなかの相関ではないでしょうか?繰り返しになりますが、結構狭い範囲での学習・予測なので(ケモタイプが絞られてる)、新たなケモタイプについて予測値を出そうとすれば大外れすると思います。でも、簡単に予測値を出すことができて結構楽しいです。
さて、キャプチャのplotですが、はっきり言ってダサいですね。綺麗じゃないというか、こなれてないというか…。
そうなんです。個人的な意見ですが、KNIMEは可視化に関してはあまり強くはないと思います。情報は得られるがかっこよくない。ですので、私は整形したファイルをspotfireなどに読ませて可視化部分を担当させています。
spotfireは有償なので、オープンソースでかっこよく描画ができるものがあるといいなーと思っています。グラフ内でインタラクティブに動かせるやつがいいです。(ご存知の方がいれば教えてください)
今回は、雑記ということでKNIMEに関する紹介は少ない&雑でしたが、いかがでしたか?私は末端の合成化学者ですので、データ解析や機械学習などについてあまり知識はありませんが、興味はもっています。現役でベンチワークしてますので、あまり勉強する時間が取れないのですが…。
周りにあまり詳しい人もいないので、いろいろと教えてくれる人がいるといいなーと思っています。何かブログ内で間違ったいる部分の指摘とか、色々な関連する面白い技術とか、コメントお待ちしていますので、もし気が向けば宜しくお願いします。
もちろん、ただの感想でも結構です。
ではまた次回!
