KNIMEで化合物をクラスタリング&可視化してみよう
みなさん、こんにちは。
私は急な気温の変化と、ブタクサ?花粉で体調がイマイチです。
心はあまり元気でないので、せめて体は元気で過ごしたいものです。
さて、今回は何について書こうかなーと考えていたのですが、前回、ChEMBLからとってきた情報を使ってクラスタリングでもしてみようかなと思います。

上記のように、前回はNS5Bポリメラーゼ阻害剤関する1697化合物ついて、文献名と活性値を持つテーブルを作成しました。これを構造ベースでクラスタリングしていきます。結果をわかりやすくするために、記載されている化合物数が多い文献上位5件に関して作業をしていきます。
全体のフローとしてはこんな感じです↓

それではスタートです!
化合物数上位5文献の絞り込み
当然ですが、そんなノードはありません。どうすれば自分のやりたいことができるかをイメージしなくてはなりません。複雑な処理でなければ、これまでに過去記事で紹介したノードを組み合わせれば結構いろんなことができると思います。
さて、今回の場合はまずValue Counterを使って、文献名の重複をカウントします。この結果ゲットできるのは、各文献に何化合物記載されているか?という内容と同義です。

この結果のcountを大きい順にソートします。
Sorter
既に紹介済みかと思っていましたが、まだでした。名前通りのノードです。

ソートしたいカラムを指定して、昇順 or 降順のどちらにするか選びます。new columnsをクリックすると、優先順位が2番目のカラムを指定してソートできます。3番目、4番目と項目を増やしていくことができます。今回は上記のように設定し、続くRow filterで最初から5行のデータを抽出します。

ここまでできたら、Reference Row Splitterを使って全体のデータから、下のルートで作成した文献名をもつものだけを抽出します。

化合物のクラスタリングをするという目的には関係無い内容でしたが、ここまでで前処理はおしまいです。
いよいよクラスタリングを実施します。
今回はECFP4のtanimoto similarityで分類します。*1
まずはFingerprintsノードをつかってECFP4を吐き出させます。*2
Distance Matrix Caluclate
各行のデータ間の距離を総当たりで計算してくれます。

色んな手法の距離を計算してくれますが、今回は前述の通りtanimotoを選びます。すると、Fingerprintsノードにより吐き出された結果のカラム(bit vector)が緑枠内に入ってくるはずです。上記のような設定で実行してみます。

Distanceカラムが新たに作成されました。ちょっとわかりにくい書き方ですが、総当たりの距離が格納されています。【総当たり対戦表の、対角線で区切った片側の三角部分】のようなイメージです。この距離の近いもの同士のグループを作ってやることで類似化合物群をまとめていきます(クラスタリング)。
k-Medoids
k-medoid法によるクラスタリングを実行します。medoid(最もクラスタの中心付近に位置するオブジェクト)をもとにして評価する方法です。*3

設定画面では、先ほど作成したDistanceカラムが"Distance matrix column"として指定されていることを確認します。そして【何個のクラスターに分けるか?】をPartition count (k)の値として指定します。今回は5つのクラスターに分けてみます。

実行後、clustered inputを確認すると"Cluster"というカラムが新たに作成されていることがわかります。この中身には割り当てられたクラスターのmedoidのRowIDが記載されています。*4

Medoids and Sizeを確認すると、medoid(今回は5つ)と、同じクラスターに含まれる化合物数がpartition sizeというカラムを見ることで把握できます。
実質、ノード3個で化合物のクラスタリングは可能です。結構簡単だと思いませんか?もし機会があればお試しください。
クラスターの可視化
ここからはおまけです。
今回紹介したクラスタリングは、谷本距離の近いもの同士をまとめるというものでした。ということは、途中で作成したDistance Matrixの次元を2次元や3次元に削減してあげれば可視化できそうですよね。ということでMDSを使って可視化していこうと思います。*5
MDS (DistMatrix)
いい感じの名前のノードがあります。distance matrixカラムの次元を落としてくれます。

今回は3次元に落としてみます。Output dimensionsを3にします。Distance matrix columnが間違いなく指定されていることも確認してください。

実行すると、MDS Col1~3の3つのカラムが新たに作成されます。distance matrixが3次元で表されたということです。これをプロットしてみます。
2D/3D Scatterplot
名前のままです。2次元・3次元の散布図を描いてくれます。

表示に必要なカラムを緑枠内に残して、実行してください。
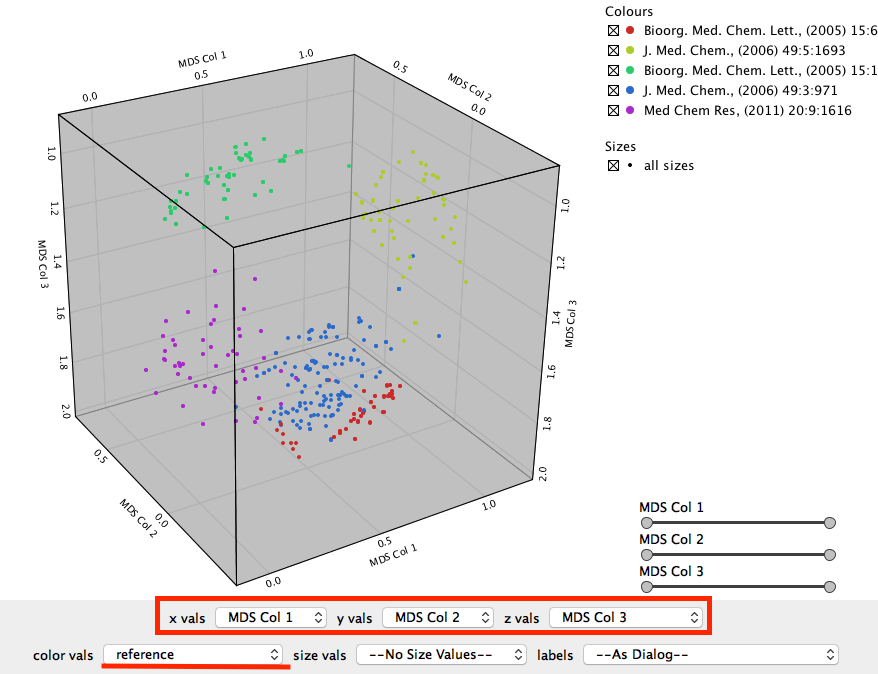
実行後、3つの軸をそれぞれ先ほど作成したMDS Col1~3に設定します。次に色ですが、まずはreference(文献名)にしてみました(このために今回の記事では文献を5件に絞りました)。
次に色をClusterに変えてみます。これは先ほどk-medoidで作成したクラスターです。
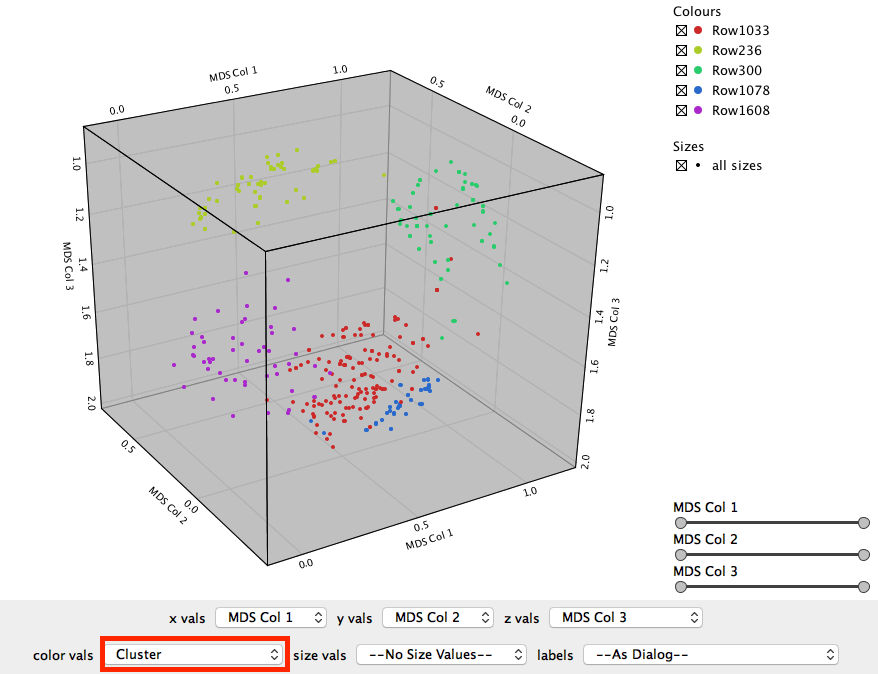
5つのグループに分かれていて、先ほど文献名で色をつけた場合とほぼ同じですね(色自体は対応してませんよ!分かれ方が同じという意味です。)。つまり、各文献で紹介されている化合物群はそれぞれECFP4で違うケモタイプであると分類することができているということがわかります。もし「文献が違うのに、fingerprintで分類すると同じ」ようなものがあれば、同じ研究グループの論文かもしれませんね。はたまたパクりかも…!?
自分のグループの化合物は、どのグループと似ているか?とかも視覚的にわかるようになると思います。そうすると参考になりやすい例とかも取得しやすくなるかもしれません。なるべくなら、先行する化合物が無い土俵で戦いたいものですが。
などなど思いつきを書きました。
次回はメドケム関連じゃなくて、もっと一般的な使い道のあるノードを何か紹介しようと思っています。
他の誰かのブログにコメントとかしたことない私が言うのおかしな気がしますが、
- 興味がわいたよー
- (既に使っていて)他にもこんな活用法があるよー
- これは間違ってるよ
などなど、コメントいただけると励みになります。
それでは、また次回!
*1:何それ?聞いたことないけど?という方は、この辺りは全部書くと長いので、 分子類似性 - Wikipediaなどを入り口にしてググっていくとたくさん記事が見つかると思います。丸投げでごめんなさい
*2:いつも、とても勉強になる内容が更新されているブログの紹介です。リンク先の記事にはE3FPについて、元文献と共にRDKit(python)での使い方も書かれています。
3d conformer fingerprint calculation using RDKit # RDKitiwatobipen.wordpress.com
こういった新しい手法もKNIMEノードではやく対応してくれると、私のようなへっぽこは助かるんですがね。…pythonも勉強しなさいってことですね。
*3:KNIMEでは他にもk-mean法(クラスターに含まれるオブジェクトの重心をもとに評価する方法)ノードもあります。
*4:クラスター前のデータのRowIDを化合物のIDなどにしておくとわかりやすいかもしれません
*5:MDSについては多次元尺度構成法 - Wikipediaなどを入り口にして、ググってみてください。これも丸投げでごめんなさい。
